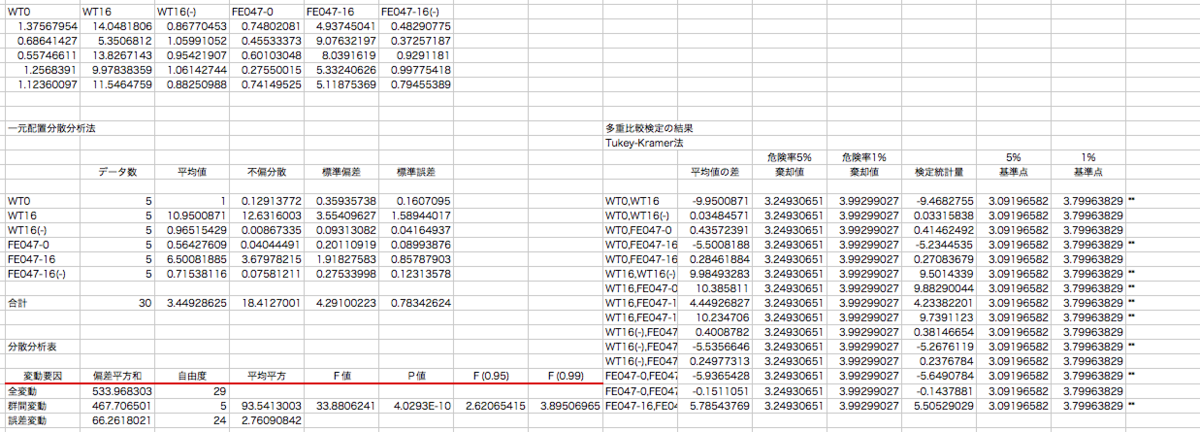
Student's T testはexcelでも特に難しくない。標準関数でできるからね。それに比べて多重検定はアドインとか手計算とか結構面倒なので、これをwebアプリでチョチョイとできるとすごくいい。というわけでまずはexcelで実施した場合。
https://www.amazon.co.jp/dp/4434211625/ref=cm_sw_em_r_mt_dp_U_y7YcEb6ERHBNK
この本(正確にはこの第3版)の付録のアドインで解析した結果。
検定統計量の絶対値が水準点より大きいと有意差があると判定される(アスタリスクのついているところ)。
さてこういうのをpythonで行うにはstatsmodels.stats.multicompモジュールの pairwise_tukeyhsd (Tukey's Honest Significant Difference)関数を用いるらしい。
$ python3
Python 3.6.5 (default, Apr 25 2018, 14:26:36)
[GCC 4.2.1 Compatible Apple LLVM 9.0.0 (clang-900.0.39.2)] on darwin
Type "help", "copyright", "credits" or "license" for more information.
>>> from statsmodels.stats.multicomp import pairwise_tukeyhsd
>>> import numpy as np
>>> A = np.array([1.375679545,0.686414272,0.557466114,1.256839102,1.123600968])
>>> B = np.array([14.04818061,5.350681195,13.82671427,9.978383591,11.54647591])
>>> C = np.array([0.867704526,1.059910525,0.954219068,1.061427443,0.882509883])
>>> D = np.array([0.748020814,0.455333729,0.601030484,0.275500152,0.741495253])
>>> E = np.array([4.937450411,9.076321974,8.039161896,5.332406264,5.118753692])
>>> F = np.array([0.482907748,0.372571868,0.929118102,0.997754185,0.79455389])
>>> data_arr = np.hstack( (A,B,C,D,E,F) )
>>> ind_arr = np.repeat(list('ABCDEF'),len(A))
>>> print(pairwise_tukeyhsd(data_arr,ind_arr))
Multiple Comparison of Means - Tukey HSD, FWER=0.05
=====================================================
group1 group2 meandiff p-adj lower upper reject
-----------------------------------------------------
A B 9.9501 0.001 6.7007 13.1995 True
A C -0.0348 0.9 -3.2843 3.2146 False
A D -0.4357 0.9 -3.6851 2.8137 False
A E 5.5008 0.001 2.2514 8.7502 True
A F -0.2846 0.9 -3.534 2.9648 False
B C -9.9849 0.001 -13.2344 -6.7355 True
B D -10.3858 0.001 -13.6352 -7.1364 True
B E -4.4493 0.0035 -7.6987 -1.1999 True
B F -10.2347 0.001 -13.4841 -6.9853 True
C D -0.4009 0.9 -3.6503 2.8485 False
C E 5.5357 0.001 2.2862 8.7851 True
C F -0.2498 0.9 -3.4992 2.9996 False
D E 5.9365 0.001 2.6871 9.186 True
D F 0.1511 0.9 -3.0983 3.4005 False
E F -5.7854 0.001 -9.0349 -2.536 True
-----------------------------------------------------ほうほう、それっぽい結果になった。5%水準で、2つの間に差がないという仮説が棄却されるとTrueなので、上のexcelの結果と完全に一致したと言える。1%水準でどうなるのかはどういうオプションを付けるのだろうか?
あと、アプリ化するにはどういう方向で設計しようか。
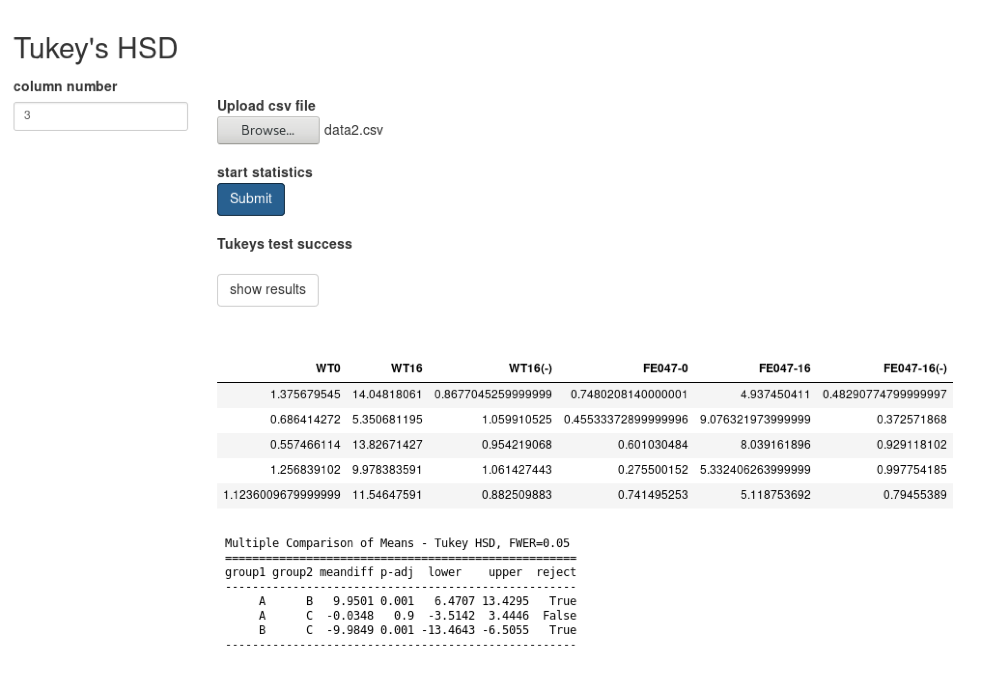
比較する項目数をどう処理しようか考え中。
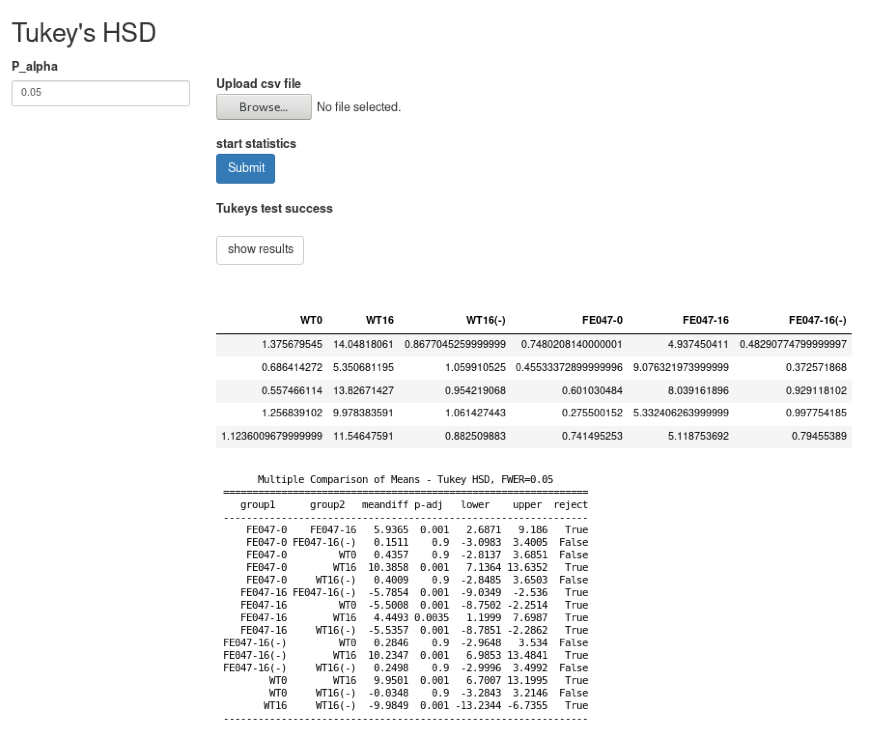
for ループで回すとよいのだろうけど、ちょっと泥臭くif分岐でごまかした。多重比較と言っても12以上も比較することはないだろうからよかろう。
@app.route('/tukey', methods=['GET', 'POST'])
def tukey_index():
user = g.user.name
form=StatForm()
dir = "./user/" + user + "/tukey"
if os.path.exists(dir):
shutil.rmtree(dir)
if not os.path.exists(dir):
os.makedirs(dir)
input = dir + "/input.txt"
output = dir + "/results.txt"
if request.method == 'POST':
if 'file' not in request.files:
flash('No file part')
return redirect(request.url)
file = request.files['file']
if file.filename == '':
flash('No selected file')
return redirect(request.url)
if file and allowed_file(file.filename):
file.save(input)
# if form.col1.data:
# col1 = form.col1
# if form.col2.data:
# col2 = form.col2
df = pd.read_csv(input)
header = df.columns
record = df.values.tolist()
# dfs1 = df.iloc[:, 0]
# dfs2 = df.iloc[:, 1]
# dfs3 = df.iloc[:, 2]
data_0 = np.array(df.iloc[:, 0].values.tolist())
data_1 = np.array(df.iloc[:, 1].values.tolist())
data_2 = np.array(df.iloc[:, 2].values.tolist())
# if form.n_samp.data == 3:
data_arr = np.hstack((data_0,data_1,data_2))
# ind_arr = np.repeat(list('abc'),len(data_0))
ind_arr = np.array([])
ind_arr = np.append(ind_arr, np.repeat(header[0],len(data_0)))
ind_arr = np.append(ind_arr, np.repeat(header[1],len(data_1)))
ind_arr = np.append(ind_arr, np.repeat(header[2],len(data_2)))
count = len(header)-3
if count > 0:
count = count -1
data_3 = np.array(df.iloc[:, 3].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3))
ind_arr = np.append(ind_arr, np.repeat(header[3],len(data_3)))
if count > 0:
count = count -1
data_4 = np.array(df.iloc[:, 4].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4))
ind_arr = np.append(ind_arr, np.repeat(header[4],len(data_4)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 5].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5))
ind_arr = np.append(ind_arr, np.repeat(header[5],len(data_5)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 6].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5,data_6))
ind_arr = np.append(ind_arr, np.repeat(header[6],len(data_6)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 7].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5,data_6,data_7))
ind_arr = np.append(ind_arr, np.repeat(header[7],len(data_7)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 8].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5,data_6,data_7,data_8))
ind_arr = np.append(ind_arr, np.repeat(header[8],len(data_8)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 9].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5,data_6,data_7,data_8,data_9))
ind_arr = np.append(ind_arr, np.repeat(header[9],len(data_9)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 10].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5,data_6,data_7,data_8,data_9,data_10))
ind_arr = np.append(ind_arr, np.repeat(header[10],len(data_10)))
if count > 0:
count = count -1
data_5 = np.array(df.iloc[:, 11].values.tolist())
data_arr = np.hstack((data_0,data_1,data_2,data_3,data_4,data_5,data_6,data_7,data_8,data_9,data_10,data_11))
ind_arr = np.append(ind_arr, np.repeat(header[11],len(data_11)))
results = str(pairwise_tukeyhsd(data_arr,ind_arr, alpha=form.alpha.data))
with open(output, mode='w') as f:
f.write(results)
link='Tukeys test success<br><br><a href="/tukey/dl/" target="info2" class="btn btn-default">show results</a>'
return render_template('/tools/tukey.html', form=form, link=link, header=header, record=record)
return render_template('/tools/tukey.html', form=form)
@app.route('/tukey/dl/')
def dl_tukey_index():
user = g.user.name
dir = "./user/" + user + "/tukey"
if os.path.exists(dir):
result = dir +"/results.txt"
f = open(result, 'r')
return Response(f.read(), mimetype='text/plain')













