相変わらずいろいろ画策中。
やっぱり何が面倒ってab1ファイルを開いて2つ重なったピークを分離するところなわけで。
ピークコールの自動化ができるととても楽ちんになる。
BiopythonモジュールでもSeqデータを見られるらしい。
from Bio import SeqIO
from Bio.SeqIO import AbiIO
test = SeqIO.read('/Volumes/Documents/works/testseq.ab1', 'abi')
print(test.annotations['abif_raw']['DATA1'])
(-7, 6, -7, -4, -8, 1, -4, -1, -3, 0, -2, -12, -3, -4, 6, 3, -4, -4, -1, 2, -3, -15, -6, -5, -8, -4, 1, -14, -8, -4, -3, -7, -6, -8, -3, 0, -3, -6, -12, -10, 0, 0, -15, -7, -14, -8, 0, -10, -2, -13, -3, -10, -8, -8, -14, -3, -9, -7, -10, -7, -8, -5, -5, -11, 0, -7, -1, -8, -6, -6, -4, -2, -13, -10, -5, -6, -9, -8, -4, -6, -19, -11, -11, -8, -6, 0, -2, -6, -8, -13, -5, -8, -4, -7, -6, -4, -10, -4,
・・・・・・
4, 488, 501, 529, 523, 530, 531, 550, 566, 577, 583, 579, 592, 577, 584, 593, 610, 597, 631, 633, 612, 612, 623, 645, 660, 649, 665, 688, 666, 640, 652, 658, 652, 660, 650, 635, 654, 667, 616, 609, 600, 588, 568, 550, 517, 524, 517, 510, 494, 470, 457, 439, 417, 408, 401, 396, 360, 334, 334, 317, 306, 285, 270, 241, 255, 230, 224, 203, 215, 188, 191, 192, 172, 151, 148, 146, 150, 137, 151, 144, 134, 136, 150, 145, 124, 132, 137, 145, 138, 143, 132, 126, 140, 134)こんな感じにプロットを出せるのね。
ということはこれを4塩基で重ねていけば、ヘテロで重なったsecondary peakも取れるんだろうか。
とりあえずtestオブジェクトのアトリビュートを確認すると
>>> dir(test) ['__add__', '__bool__', '__class__', '__contains__', '__delattr__', '__dict__', '__dir__', '__doc__', '__eq__', '__format__', '__ge__', '__getattribute__', '__getitem__', '__gt__', '__hash__', '__init__', '__init_subclass__', '__iter__', '__le__', '__le___', '__len__', '__lt__', '__module__', '__ne__', '__new__', '__nonzero__', '__radd__', '__reduce__', '__reduce_ex__', '__repr__', '__setattr__', '__sizeof__', '__str__', '__subclasshook__', '__weakref__', '_per_letter_annotations', '_seq', '_set_per_letter_annotations', '_set_seq', 'annotations', 'dbxrefs', 'description', 'features', 'format', 'id', 'letter_annotations', 'lower', 'name', 'reverse_complement', 'seq', 'upper']
こんな感じ。
>>> test.id
'1I1_F'
>>> test.name
'1I1_F_P1815443_047'
>>> test.seq
Seq('CNGNNNCGAGTCTTTGATGCAGTTGCGCTCGAGGCCATTANGTTGAGAGCACGT...NNN', IUPACUnambiguousDNA())
>>> test.reverse_complement()
SeqRecord(seq=Seq('NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN...CNG', IUPACUnambiguousDNA()), id='<unknown id>', name='<unknown name>', description='<unknown description>', dbxrefs=[])( ちなみにここで見ているシークエンスファイルはネットに落ちていたもので何かは知らない)
さらにannotationsは
>>> test.annotations.keys()
dict_keys(['sample_well', 'dye', 'polymer', 'machine_model', 'run_start', 'run_finish', 'abif_raw'])
>>> for k,v in test.annotations.items():
... print('key:'+k+' , values:'+v)
...
key:sample_well , values:A5
key:dye , values:Z-BigDyeV3
key:polymer , values:POP7
key:machine_model , values:3730
key:run_start , values:2018-05-25 18:23:59
key:run_finish , values:2018-05-25 20:38:32
Traceback (most recent call last):
File "<stdin>", line 2, in <module>
TypeError: must be str, not dictabif_rawキーに対するvalueには
'DATA1': 'Channel 1 raw data',
'DATA2': 'Channel 2 raw data',
'DATA3': 'Channel 3 raw data',
'DATA4': 'Channel 4 raw data',
'DATA5': 'Short Array holding measured volts/10 (EP voltage) during run',
'DATA6': 'Short Array holding measured milliAmps trace (EP current) during run',
'DATA7': 'Short Array holding measured milliWatts trace (Laser EP Power) during run',
'DATA8': 'Short Array holding measured oven Temperature (polymer temperature) trace during run',
'DATA9': 'Channel 9 processed data',
'DATA10': 'Channel 10 processed data',
'DATA11': 'Channel 11 processed data',
'DATA12': 'Channel 12 processed data',となっているようで、
>>> print(test.annotations['abif_raw']['DATA9'][100]) 196
というふうに数値を取り出せる。
ピークを抽出するのはどうする?
9~12のデータの並び順は
>>> print(test.annotations['abif_raw']['FWO_1']) GATC
からGATCの順のようだ。
と、いろいろ調べてきたが、結局2番めに高いピークをひろうにはすでにあるab1 fileではどうにもならないようだ。
ベースコールを行うphredで-dオプションを付けて再解析する必要があるらしい。
しかしPhredのダウンロードサイトが閉じているようで、入手ができそうにない。
Mac,Windowsのデスクトップアプリはあるようなのだけど、それだと自前のwebアプリにビルトインできないからなあ。
やっぱりコマンドラインで動くUnix版がほしいところ。
というわけで代わりにTraceTunerを試してみる。
sourceforge.net
これまたとっつきにくいアプリケーションの雰囲気を漂わせている。CVSだけど、気にせずsrc/に入ってmake
めちゃめちゃワーニングが出るな。
とりあえず、
$ cd ../rel/Linux
$ ./ttuner -h
-h (Help) This message
-Q (Quiet) Turn off status messages
-V (Verbose) Output more status messages
-nocall Disable base recalling and just use the original
called bases read from the input sample file
-recalln Disable adding bases to or deleting from the
original called sequence. Only recall Ns
-het Call call hetezygotes
-mix Call mixed bases
-min_ratio <ratio> Override the default threshold ratio of heights of
-trim_window <size> Set the trimming window size for averaging quality
to the specified value. The default is 10.
-trim_threshold <qv> Set the average quality value used in trimming to
-C <consensusfile> Specify the name of the FASTA file which contains
the consensus sequence
-edited_bases Start base recalling from the ABI's edited bases
-t <table> Use specified lookup table. This option overrides
the default (automatic choice of the lookup table)
as well as the options -3700pop5, -3700pop6, -3100,
and -mbace. To get a message showing
which table was used, specify -V option
-3730 Use the built-in ABI 3730-pop7 lookup table
-3700pop5 Use the built-in ABI 3700-pop5 lookup table
-3700pop6 Use the built-in ABI 3700-pop6 lookup table
-3100 Use the built-in ABI 3100-pop6 lookup table
-mbace Use the built-in MegaBACE lookup table
-c Output SCF file(s), in the current directory
-cd <dir> Output SCF file(s), in the specified directory
-cv3 Use version 3 for output SCF file. Default is
version 2.
-o <dir> Output multi-fasta files of bases (tt.seq),
their locations (tt.pos), quality values (tt.qual)
and status reports (tt.status) to directory <dir>
-p Output .phd.1 file(s), in the current directory
-pd <dir> Output .phd.1 file(s), in the specified directory
-q Output .qual file(s), in the current directory
-qa <file> Append .qual file(s) to <file>
-qd <dir> Output .qual file(s), in the specified directory
-s Output .seq file(s) in FASTA format, in the current
directory
-sa <file> Append .seq file(s) in FASTA format to <file>
-sd <dir> Output .seq file(s) in FASTA format, in the specified
directory
-qr <file> Output a quality report that gives data for a
histogram on the number of reads with quality
values >= 20, to the specified file
-if <file> Read the input sample filenames from the specified
file
-id <dir> Read the input sample files from specified directory
-tab Call heterozygotes or mixed bases and output .tab
file(s) in the current directory
-tabd <dir> Call mixed bases and output .tab file(s), in the
specified directory
-tal Output .tal file(s),in the current directory
-tald <dir> Output .tal file(s),in the specified directory
-hpr Output a homopolymer runs file in current directory
-hprd <dir> Output a homopolymer runs file(s),in the specified directory
-d Output .poly file(s),in the current directory
-dd <dir> Output .poly file(s),in the specified directory
-ipd <dir> Input the original bases and peak locations from a
.phd file in the specified directory.
さてオプションがいっぱいあるが
$ /Applications/tracetuner_3.0.6beta/rel/Linux/ttuner -p ./1I1_F_P1815443_047.ab1
これが最も基本のオプションだろうか。
BEGIN_SEQUENCE 1I1_F_P1815443_047.ab1 BEGIN_COMMENT CHROMAT_FILE: 1I1_F_P1815443_047.ab1 ABI_THUMBPRINT: 0 PHRED_VERSION: TT_3.0.4beta CALL_METHOD: ttuner QUALITY_LEVELS: 45 TIME: Sat Jun 29 21:04:35 2019 TRACE_ARRAY_MIN_INDEX: 0 TRACE_ARRAY_MAX_INDEX: 14037 TRIM: 16 622 0.010000 CHEM: unknown DYE: big END_COMMENT BEGIN_DNA c 14 114 g 16 118 g 13 135 t 17 138 c 7 154 t 7 168 c 13 176 g 11 183 a 13 199 g 18 203 t 17 219 c 17 223 t 20 235 t 14 245 t 14 255 g 14 259 a 15 276 t 15 289 g 16 295 c 16 306 a 17 319 g 24 330 t 25 346 t 26 357 g 26 364 c 25 376 g 26 387 c 25 399 t 26 414
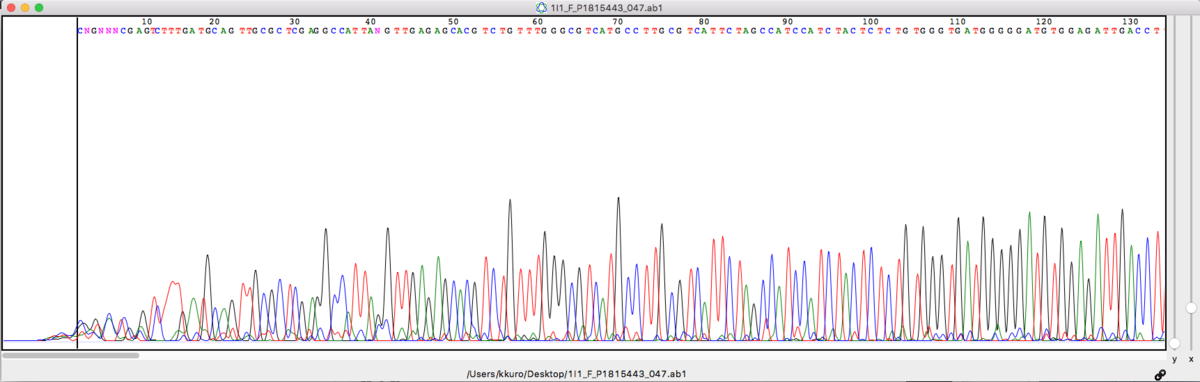
上に上げたKB basecallerで読まれたシークエンス(チャート)と比べると
CNGNNNCGAGTCTTTGATGCAGTTGCGC CGGTCTCGAGTCTTTGATGCAGTTGCGC
とまあほぼ一致している。
$ /Applications/tracetuner_3.0.6beta/rel/Linux/ttuner -mix -tab ./1I1_F_P1815443_047.ab1
こんな感じに投げてみると
# CHROMAT_FILE: 1I1_F_P1815443_047.ab1 # SOFTWARE_VERSION: TT_3.0.4beta # NUM_ABC: 236 # NUM_SUBSTITUTIONS: 236 # NUM_DELETIONS: 0 # base2 qv2 pos2 ind2 Y 11 115 0 T 7 116 0 S 13 133 2 C 7 131 2 A 7 154 4 C 7 154 4 M 10 176 6 A 7 176 6 K 8 187 7 T 7 192 7 M 12 198 8 C 10 198 8 W 11 254 14 T 7 255 14 R 14 262 15 S 9 260 15 A 9 265 15 C 11 261 15 M 9 277 16 C 7 279 16 W 11 290 17 A 7 292 17 W 16 319 20 T 9 319 20 T 9 332 21 G 25 330 21 W 18 356 23 A 11 355 23 Y 11 374 25 T 9 373 25 K 24 385 26 T 9 384 26 W 23 412 28 A 9 411 28 ・・・・・・・・・・ ・・・・・・・・・・
こういうファイルが出来上がるわけだが、これでいいのかな?
CNGNNNCGAGTCTTTGATGCAGTTGCGC CGGTCTCGAGTCTTTGATGCAGTTGCGC YTSCACMAKTMCWTRSACMCWAWTTGWAY
なんかおかしいね。
$ /Applications/tracetuner_3.0.6beta/rel/Linux/ttuner -min_ratio 0.1 -d ./1I1_F_P1815443_047.ab1
こうすると、最も高いピークに加えて10%以上の高さのピークを次に選んでくれそう
1I1_F_P1815443_047.ab1 1.0 1.0 1.0 1.0 1.0 C 114 2990.000000 1.207398 N -1 -1.000000 -1.000000 51.000000 196.000000 155.000000 79.000000 G 118 3210.000000 1.296236 T 115 912.500000 0.368478 62.000000 140.000000 170.000000 72.000000 G 135 2444.500000 0.987118 C 132 935.500000 0.377766 59.000000 61.000000 208.000000 133.000000 T 138 2012.500000 0.812672 N -1 -1.000000 -1.000000 37.000000 35.000000 173.000000 154.000000 C 154 3376.500000 1.363471 A 154 3609.500000 1.457559 215.000000 265.000000 0.000000 0.000000 T 168 1846.500000 0.745639 N -1 -1.000000 -1.000000 25.000000 76.000000 0.000000 133.000000 C 176 2879.000000 1.162575 A 176 268.500000 0.108424 29.000000 224.000000 17.000000 102.000000 G 183 1223.000000 0.493862 N -1 -1.000000 -1.000000 5.000000 85.000000 122.000000 5.000000 A 199 1633.000000 0.659425 C 198 852.500000 0.344250 136.000000 89.000000 199.000000 0.000000 G 203 3149.000000 1.271604 N -1 -1.000000 -1.000000 110.000000 44.000000 286.000000 0.000000 T 219 4653.500000 1.879139 N -1 -1.000000 -1.000000 0.000000 178.000000 4.000000 425.000000 C 223 3368.000000 1.274430 N -1 -1.000000 -1.000000 0.000000 329.000000 0.000000 299.000000 T 235 2353.000000 0.885069 N -1 -1.000000 -1.000000 0.000000 0.000000 0.000000 351.000000 T 245 7217.500000 2.724202 N -1 -1.000000 -1.000000 22.000000 0.000000 0.000000 574.000000 T 255 5279.000000 1.665352 A 252 912.000000 0.287706 94.000000 2.000000 132.000000 536.000000 G 259 2757.000000 0.820499 A 262 726.500000 0.216211 77.000000 36.000000 273.000000 317.000000 A 276 6011.000000 1.741713 C 280 659.000000 0.190948 411.000000 39.000000 0.000000 0.000000 T 289 3652.000000 0.970141 N -1 -1.000000 -1.000000 101.000000 3.000000 233.000000 404.000000 G 295 8538.000000 2.130612 A 293 841.500000 0.209992 84.000000 0.000000 831.000000 144.000000 C 306 3019.000000 0.642641 N -1 -1.000000 -1.000000 14.000000 315.000000 11.000000 8.000000 A 319 4293.000000 0.916368 T 318 794.500000 0.169591 312.000000 0.000000 9.000000 73.000000 G 330 4400.000000 0.946491 T 332 857.000000 0.184351 23.000000 0.000000 391.000000 101.000000 T 346 5086.500000 1.070403 N -1 -1.000000 -1.000000 14.000000 14.000000 1.000000 550.000000 T 357 4582.000000 0.911786 C 352 601.000000 0.119595 46.000000 17.000000 83.000000 510.000000 G 364 6482.000000 1.361264 N -1 -1.000000 -1.000000 2.000000 0.000000 679.000000 62.000000 C 376 5179.000000 1.060825 T 373 475.000000 0.097295 0.000000 455.000000 0.000000 58.000000 G 387 4755.000000 0.927941 T 383 813.000000 0.158657 0.000000 90.000000 488.000000 46.000000 C 399 6513.500000 1.303052 N -1 -1.000000 -1.000000 0.000000 592.000000 0.000000 0.000000 T 414 4645.000000 0.878936 A 412 989.000000 0.187140 94.000000 73.000000 0.000000 544.000000 C 421 4935.500000 1.008171 A 421 501.000000 0.102339 57.000000 513.000000 0.000000 66.000000
これかな。
$ /Applications/tracetuner_3.0.6beta/rel/Linux/ttuner -mix -s ./1I1_F_P1815443_047.ab1
こっちでもいいかも
>1I1_F_P1815443_047.ab1 1195 16 579 CGGTMTCGAGTCTTAGATGCAKTTGCGCTCGASGCCATTASSTTGAGAGC ACGTCTGTTTGGGCGTCATGCCTTGCGTCATTCTAGCCATCCATCTACTC TCTGTGGGTGATGGGGGATGTGGAGATTGACCTTCCGTGCTTTAATTGTA
なお
R A or G Y C or T S G or C W A or T K G or T M A or C B C or G or T D A or G or T H A or C or T V A or C or G N any base