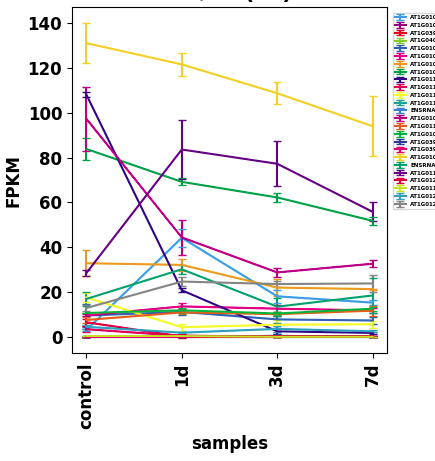
marplotlibは複数の要素をグラフ表示すると、自動的に色を分けてくれるわけだが、これが10色しか用意されていない。まあ実際10本以上折れ線が重なっていたらかなり見にくいことになるのは確かなのだが、それでもたくさんの色で表現したいこともあるわけで。そこでこの色の自動振り当てをどうにかする方法を考えた。
color_codes={3:'#E60012',7:'#F39800',11:'#FFF100',4:'#8FC31F',8:'#009944',12:'#009E96',1:'#00A0E9',5:'#0068B7',9:'#1D2088',2:'#920783',6:'#E4007F',10:'#E5004F',15:'#EB6100',19:'#FCC800',23:'#CFDB00',16:'#22AC38',20:'#009B6B',24:'#00A0C1',13:'#0086D1',17:'#00479D',21:'#601986',14:'#BE0081',18:'#E5006A',22:'#E60033',25:'#848484',0:'#000000'}まずはこういうカラーチャートに番号を振っておく。番号が順番どおりでないのはグラフに表示させる順番で近い色が隣り合わせにならないように調整したため。
c_code = 0
#と初期化しておいて複数の要素を描かせるためにforループを回す。
for y in q1:
y0 = [y.data1, y.data2, y.data3]
y2 = [y.data10,y.data11,y.data12]
y3 = [y.data13,y.data14,y.data15]
y5 = [y.data19,y.data20,y.data21]
y0m = statistics.mean(y0)
y2m = statistics.mean(y2)
y3m = statistics.mean(y3)
y5m = statistics.mean(y5)
y0d = statistics.stdev(y0)
y2d = statistics.stdev(y2)
y3d = statistics.stdev(y3)
y5d = statistics.stdev(y5)
c_code1 = c_code % 26
gcolor = color_codes[c_code1]
#ここでループごとの色指定を行う。26色を用意しているので26で割ったあまりの数値で指定する。27番目で最初の色に戻るわけだ。
x_ax = np.array([0, 1, 2, 3])
y_ax = np.array([y0m,y2m,y3m,y5m], dtype=np.float16)
y_err = np.array([y0d,y2d,y3d,y5d])
ax.errorbar(x_ax, y_ax, y_err, capsize=3, linestyle=lineStyle, linewidth=lineWidth, color=gcolor, marker=mstyle, markersize=mSize, markeredgewidth=1, label = y.id)
plt.xticks([0,1,2,3], ['control','1d','3d','7d'], rotation=90, fontsize=tSize, weight='bold')
ax.legend(bbox_to_anchor=(1, 1), loc='upper left', borderaxespad=1, fontsize=lSize)
plt.xlabel('samples', fontsize=xSize, weight='bold')
plt.ylabel('FPKM', fontsize=ySize, weight='bold')
plt.tick_params(axis='y', which='major', labelsize=ytSize)
plt.tight_layout()
plt.subplots_adjust(left=0.15, right=0.6)
img_data1a = dir + "a_" + str(time.time()) + ".eps"
plt.savefig(img_data1a)
canvas = FigureCanvasAgg(fig)
buf = io.BytesIO()
canvas.print_png(buf)
data = buf.getvalue()
img_data = urllib.parse.quote(data)
#ここまででグラフを描写
c_code = c_code + 1
#これで色指定をシフトさせる。