マイクロアレイや、次世代シークエンサーによる発現データセットが大量にあると、すべての遺伝子同士の制御関係(上下関係)が描けるかもしれない。
ということで方法を模索するとネットワーク解析というものに行き着く。ところが、世の中に出ている遺伝子発現関係の論文でネットワーク解析というと大体は共発現ネットワーク(co-expression network)であるらしく、制御関係を示すネットワークではないらしい。
そういう関係性を推論するにはベイジアンネットワーク(bayesian network)解析を行うといいらしい。しかし、どうやらこいつは膨大な計算量になるらしいこともあちこちに書かれている。とりあえず、手元の計算機資源だけで小規模にでも試してみることはできないだろうかと、また、せっかくなのでpythonでライブラリがなかろうかと探してみたところ
Peblというのが見つかった。
pypi.org
ところがこいつは開発がだいぶ前に止まっていて、python3ではインストールさえさせてもらえなかった。
$ python3 -m pip install pebl
Collecting pebl
Using cached pebl-1.01.tar.gz (2.5 MB)
ERROR: Command errored out with exit status 1:
command: /home/linuxbrew/.linuxbrew/opt/python@3.8/bin/python3.8 -c 'import sys, setuptools, tokenize; sys.argv[0] = '"'"'/tmp/pip-install-3u4r784s/pebl/setup.py'"'"'; __file__='"'"'/tmp/pip-install-3u4r784s/pebl/setup.py'"'"';f=getattr(tokenize, '"'"'open'"'"', open)(__file__);code=f.read().replace('"'"'\r\n'"'"', '"'"'\n'"'"');f.close();exec(compile(code, __file__, '"'"'exec'"'"'))' egg_info --egg-base /tmp/pip-pip-egg-info-assg8ojs
cwd: /tmp/pip-install-3u4r784s/pebl/
Complete output (6 lines):
Traceback (most recent call last):
File "<string>", line 1, in <module>
File "/tmp/pip-install-3u4r784s/pebl/setup.py", line 38
except DistutilsPlatformError, x:
^
SyntaxError: invalid syntax
----------------------------------------
ERROR: Command errored out with exit status 1: python setup.py egg_info Check the logs for full command output.いちおうpython2環境では使えなくもなさそうなのだけど、MacではSegmentation Fault:11で落ちるとかいろいろ問題がありそうなので、他を当たる。
$ python
Python 2.7.13 (v2.7.13:a06454b1afa1, Dec 17 2016, 12:39:47)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)] on darwin
Type "help", "copyright", "credits" or "license" for more information.
>>> from pebl import data
>>> from pebl.learner import greedy
>>> dataset = data.fromfile("pebl-tutorial-data1.txt")
>>> dataset.discretize()
>>> learner = greedy.GreedyLearner(dataset)
>>> ex1result = learner.run()
Segmentation fault: 11bayespy
github.com
これは行けそう。とりあえずチュートリアルでもやってみるかね。
import numpy as np np.random.seed(1) data = np.random.normal(5, 10, size=(10,)) from bayespy.nodes import GaussianARD, Gamma mu = GaussianARD(0, 1e-6) tau = Gamma(1e-6, 1e-6) y = GaussianARD(mu, tau, plates=(10,)) y.observe(data) from bayespy.inference import VB Q = VB(mu, tau, y) Q.update(repeat=20) import bayespy.plot as bpplt bpplt.pyplot.subplot(2, 1, 1) bpplt.pdf(mu, np.linspace(-10, 20, num=100), color='k', name=r'\mu') bpplt.pyplot.subplot(2, 1, 2) bpplt.pdf(tau, np.linspace(1e-6, 0.08, num=100), color='k', name=r'\tau') bpplt.pyplot.tight_layout() bpplt.pyplot.show()
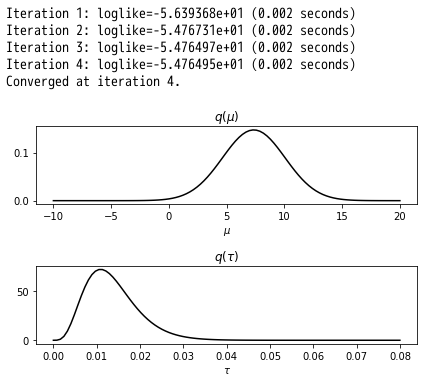
さて、これは何なんだろうね?w
なおインストールしたまま実行すると
Traceback (most recent call last):
File "bay1.py", line 13, in <module>
Q.update(repeat=20)
File "/home/linuxbrew/.linuxbrew/Cellar/python@3.8/3.8.5/lib/python3.8/site-packages/bayespy/inference/vmp/vmp.py", line 163, in update
t = time.clock()
AttributeError: module 'time' has no attribute 'clock'
[rnaseq@mn-rx1330m3 temp]$ nano bay1.py
[rnaseq@mn-rx1330m3 temp]$ python3 bay1.py
Traceback (most recent call last):
File "bay1.py", line 13, in <module>
Q.update(repeat=20)
File "/home/linuxbrew/.linuxbrew/Cellar/python@3.8/3.8.5/lib/python3.8/site-packages/bayespy/inference/vmp/vmp.py", line 163, in update
t = time.clock()
AttributeError: module 'time' has no attribute 'clock'こういうエラーが出るので
"/home/linuxbrew/.linuxbrew/Cellar/python@3.8/3.8.5/lib/python3.8/site-packages/bayespy/inference/vmp/vmp.py"
の中にあるtime.clock()をすべてtime.process_time()に書き換えてやる(python3.8以上の場合、3.7以下ならエラーは出ないみたい)
コードを読みほぐしてみる
import numpy as np np.random.seed(1) data = np.random.normal(5, 10, size=(10,))
まずNumpyでランダムなデータセットを作成しているな。
Gaussian distribution(正規分布する)平均5、SDが10な10個の数値を生成している。
from bayespy.nodes import GaussianARD, Gamma mu = GaussianARD(0, 1e-6) tau = Gamma(1e-6, 1e-6) y = GaussianARD(mu, tau, plates=(10,))
ここでmodelを設定している。平均とSDを推定するモデルね。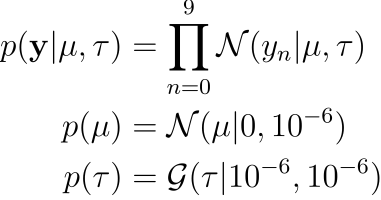
うーむガンマ分布ってなんだったっけ。
とりあえずbayespy.nodesにいろいろな分布について用意されているので、ここを自分の考えるモデルに合わせて変えてくれってことね。
さあ、モデルができたらそれに当てはめてみるよ。
y.observe(data)
事後分布を推定する。
from bayespy.inference import VB Q = VB(mu, tau, y) Q.update(repeat=20)
どうやらここで推論エンジンをつかうのだが、このライブラリではVB(variational Bayesian 変分ベイズ)エンジンしか実装していないようだ。
以下はq(µ)とq(タウ)をグラフにしている。
モデルをどうやってでっち上げるのか、ってところが問題だな。というかこれからネットワークをプロットするのは無理かも。